Feline panleukopeni virüs (FPLV) ya da feline parvo virüs (FPV) yüksek ateş, depresyon, iştah kaybı, dehidrasyon, kusma, lökopeni ve diyare ile karakterize olan evcil ve vahşi kedilerin (Mustelidea, Procyonidea ve Viverridea ailelerinde yer alan) oldukça bulaşıcı olan viral bir hastalığıdır
1,2. FPLV, Parvoviridea ailesinde Parvovirinae alt ailesinde, Protoparvovirus cinsinde, Carnivore protoparvovirus 1 taksonu içerisinde yer alır. Virüs küçük, zarfsız ve tek iplikli; yaklaşık 5kb uzunluğunda bir genoma sahip olan bir DNA virüsüdur. FPLVün kedilerde hastalık yaptığı 20. yüzyılın başlarından itibaren bilinmektedir
3. Canine parvovirus type 2 (CPV- 2) ve FLPV yaklaşık olarak %98 genom benzerliğine sahip olan, birbirleriyle yakın ilişkili iki virüstür. CPV-2 ilk olarak 1970lerin sonlarında tespit edilmiştir. Yapılan bazı çalışmalar
4-6 FPLVün mink ve tilki gibi yabani karnivorlar aracılığı ile köpek konaklarına geçtiğini göstermiştir. CPV-2 DNA virüsü olmasına rağmen, genomunda meydana gelen nispeten hızlı mutasyonlar sonucunda şimdilerde CPV-2a, CPV-2b ve CPV-2c olarak bilinen üç antijenik tipi ortaya çıkmıştır. FPLV ile CPV-2nin VP2 kapsid proteini arasında altı ya da yedi amino asit değişimi vardır
7. CPV- 2 genetik tipleri arasında da 426. amino asitteki tek nokta mutasyonu ile 3 farklı tip oluşmuştur. Bu genetik tipler (CPV-2a/2b/2c) kedilerde FPLVden klinik olarak ayırt edilemeyen gastroenteritis tablosu oluşturabilirler
8.
Parvovirusların sahip olduğu major kapsid proteini VP2, virüsün antijenik özelliklerini ortaya koyar. VP2 proteini üzerinde yer alan aminoasit rezidüleri yeni konaklara virüsun adaptasyonunda (297,300, 305, 323 ve 568 pozisyonlarında) önemli rol oynar 8-10.
FPLV klinik olarak şiddetli depresyon, abdominal ağrı, kusma, dehidrasyon, şiddetli lökopeni ve ishal ile seyreder ve sık sık ölümlere neden olur 11. FPLVün en yaygın bulaşma yolu enfekte kedilerin dışkı, idrar ve kanlarıyla duyarlı hayvanların doğrudan temasıdır. Hastalığın akut döneminde tüm vücut sıvılarıyla virüs saçılımı gerçekleşir. Günümüzde çeşitli aşılar (kedi karma aşıları ve FPL için olan aşılar) ile hastalık engellenmektedir. Ancak aşılanmayan populasyonlarda hastalık şiddetli ve yıkıcı etkilere neden olur. Hastalıktan etkin korunma yolu aşılamadır. Kediler sekiz haftalık olduktan sonra aşılama programı uygulanır 2,12. Türkiyede ticari olarak kullanılan aşılar sırasıyla, zayıflatılmış feline rhinotracheitis virüs ve calicivirus kapsayan Felocell (Zoetis) ve Johnson Snow Leopard FPLV ve zayıflatılmış feline rhinotracheitis virus kapsayan Purevax PCP (Merial)dir.
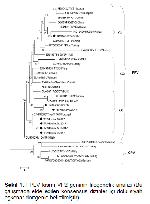
Türkiyede klinik olarak enfeksiyonun varlığı bilinmesine rağmen FPLV ile ilgili yapılan serolojik 13 ve moleküler 10,14 çalışmaların sayısı sınırlıdır.
Bu çalışmanın amacı, Mersin ilinde FPLVün varlığını düşündüren;
i-klinik bulgulu kedilerde FPLV enfeksiyonunu ortaya koymak,
ii-saptanan virüslerde kısmı VP2 geninin genetik analizini yapmak ve
iii-Türkiyede tespit edilen suşlar ve aşı suşlarıyla benzerlik/farklılığı ortaya koymaktır.